Ribosome, traduction et cancer

Objectifs
L’équipe Ribosome, Traduction et Cancer étudie le rôle émergent des ribosomes et de l’hétérogénéité des ribosomes dans le développement du cancer. Nous recherchons les altérations des ribosomes et de la machinerie de production des ribosomes qui caractérisent les cancers, avec deux objectifs : premièrement, mieux comprendre le processus de développement des tumeurs et deuxièmement, appliquer ces connaissances à l’identification de nouveaux biomarqueurs, au développement de nouvelles stratégies thérapeutiques pour traiter le cancer et à la compréhension de la réponse aux chimiothérapies. Nous nous focalisons sur les pathologies liées aux cancers du sein, du colon, du poumon et du pancréas.
Projets
Nos projets s’articulent autour de 6 thèmes, sous la direction du Dr J-J. Diaz :
- Ciblage thérapeutique des protéines externalisées. (Dr. MA. Albaret)
- Fonctions et ciblage de la Nucléoline (Pr. P. Bouvet)
- Rôle de la méthylation des ARNr dans la traduction et ciblage thérapeutique (Dr. F. Catez)
- Altération des ribosomes et de la traduction par les chimiothérapies (Dr. N. Dalla Venezia)
- Ribosome et régulations traductionnelles de la plasticité cellulaire (cellules souches embryonnaires, gliomes) (Dr. S. Durand)
- Rôle de la méthylation des ARNr dans les cancers et intérêt en clinique (Dr. V. Marcel)
Biogenèse des ribosomes et cancer
Notre groupe s’intéresse depuis longtemps aux dérégulations de la biogenèse des ribosomes qui sont associées aux pathologies et en particulier au développement tumoral et à la chimiorésistance. La biogenèse des ribosomes est un processus fondamental complexe assurant la production des ribosomes, les complexes ribonucléoprotéiques qui effectuent toutes les synthèses protéiques de la cellule. Récemment, il est apparu que tous les ribosomes d’une cellule ne sont pas identiques et que la composition d’un ribosome peut avoir un impact sur l’activité de traduction, y compris le contrôle de la qualité de la synthèse protéique.
Il est bien connu que la biogenèse des ribosomes est suractivée dans les cellules cancéreuses, pour faire face à l’augmentation de leur métabolisme et de leur prolifération. En conséquence, la morphologie des nucléoles (les domaines nucléaires où se déroule la biogénèse des ribosomes) est fortement modifiée dans les cellules cancéreuses, et constitue l’une des caractéristiques les plus distinctives des cellules tumorales. Des études génétiques menées sur divers modèles animaux ont montré que la modification de la biogenèse des ribosomes, et par conséquent de leur composition, pouvait avoir un effet bénéfique sur la survie cellulaire et donc sur le développement du cancer. Notre groupe a montré que dans les cancers, la production de ribosomes est altérée, résultant en des ribosomes de compositions distinctes, et en particulier que la méthylation des ARN ribosomiques (ARNr) est modifiée (Belin. S et al., PLoS ONE (2009) ; Marcel, V. et al., Cancer Cell (2013) ; Erales, J. et al. PNAS (2017) ; Marcel, V. et al., NAR Cancer (2020) ; Paraqindes, H. et al. Neuro. Oncol. 2023).
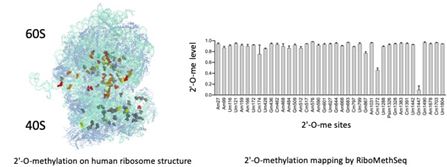
Rôle de la 2′-O-méthylation des ARN ribosomiques dans les cancers
La 2′-O-méthylation est la modification chimique la plus abondante des ARN ribosomiques (ARNr) avec 106 sites cartographiés dans les ribosomes humains. Plusieurs études, dont les nôtres, ont révélé que la 2′-O-méthylation contribue à l’activité de traduction du ribosome et a un impact sur la traduction des ARNm codant pour des protéines clés de la tumorigenèse (oncogènes, facteurs de survie…). Nous caractérisons actuellement la 2′-O-méthylation des ARN ribosomiques dans diverses pathologies et modèles de cancer pour explorer le potentiel de ces altérations dans la fonction des ribosomes et comme nouveaux biomarqueurs. Nous utilisons le RiboMeth-Seq, une nouvelle méthode basée sur le RNA-Seq qui fournit une cartographie exhaustive et quantitative de la 2′-O-méthylation dans tous les ARN ribosomiques (Erales, J. et al. PNAS (2017) ; Marcel, V. et al, NAR Cancer (2020)).
Nos projets actuels comprennent l’étude du rôle des sites variables de 2′-O-méthylation sur la fonction des ribosomes, la régulation de la traduction et le phénotype cellulaire.
Rôle des gènes de la biogenèse des ribosomes Fibrillarine et Nucléoline dans la biologie du cancer
Il est établi depuis longtemps que la biogenèse des ribosomes est dérégulée dans les cancers, ce qui contribue à l’altération quantitative et qualitative de la population de ribosomes dans les cellules cancéreuses. Dans les cellules normales, la biogenèse des ribosomes est finement régulées par de nombreuses voies de signalisation pour répondre au besoin de la cellule saine. Dans les cellules tumorales, ces voies de signalisation sont très souvent altérées et correspondent préférentiellement aux voies oncogéniques et tumeur suppressives qui induisent une dérégulation de l’ARN Pol I et donc de la biogenèse des ribosomes. En revanche, nous ne savons pas comment les étapes de maturation et d’assemblage des ribosomes contribuent à la production de ribosomes spécifiques du cancer. La fibrillarine (FBL) et la nucléoline (NCL) sont deux des facteurs les plus importants de la biogenèse des ribosomes et contrôlent à la fois la quantité et la qualité de la population de ribosomes produits. La FBL est la 2′-O-méthyltransférase des ARNr, mais elle est également nécessaire au clivage des pré-ARNr en ARNr matures et stimule l’activité de l’ARN Pol I. La NCL est nécessaire à l’activité de l’ARN Pol I, à la maturation du pré-ARN et contribue à l’assemblage des ribosomes.
Nous explorons la contribution et les rôles mécanistiques de ces deux facteurs dans plusieurs pathologies, notamment le cancer du sein, la leucémie et les cancers du pancréas (Belin. S et al, PLoS ONE (2009) ; Marcel, V. et al, Cancer Cell (2013), Marcel, V. et al PLoS ONE (2017), Nguyen Van Long F, et al Cancers, (2018), Gilles, M et al. Cancer Res. 2016, Kabirian-Dehkordi S, et al Nanomédecine (2019) ; Nguyen Van Long F, et al. BMC Cancer 2022).
Hétérogénéité des ribosomes et chimiothérapie
Notre équipe explore comment la variabilité de composition des ribosomes contribue à la résistance et au mode d’action des thérapies non ciblées et ciblées, dans le cancer du colon et du poumon.
Nous nous sommes récemment concentrés sur le 5-Fluorouracil (5-FU), la thérapie anticancer la plus administrée dans le monde. Le 5-FU a d’abord été utilisé pour son activité anti-proliférative par le biais d’effets délétères sur l’ADN. Ce n’est que récemment que nous nous sommes intéressés à l’incorporation des métabolites du 5-FU dans les ARN cellulaires. Alors que l’incorporation du 5-FU dans l’ARN est jusqu’à 15 000 fois plus élevée que dans l’ADN, les conséquences sur le fonctionnement de l’ARN restent mal comprises. Cependant, nous avons montré que le 5-FU induit une reprogrammation de la traduction par un mécanisme moléculaire qui reste à élucider. Nous avons récemment caractérisé que le 5-FU altère la biogenèse des ribosomes et leur composition (Therizols, G. et al. Nat. Commun. (2022) ; Chalabi-Dchar, M., NAR Cancer (2021)). Nous explorons comment ces modifications contribuent à la régulation de la traduction des cellules traitées et à leur phénotype.
Savoir-faire
Nous développons et exploitons un ensemble de technologies et méthodologies complémentaires pour décrire au niveau moléculaire et cellulaire chaque étape de la biogenèse des ribosomes à la fois dans des modèles cancéreux pertinents (lignées cellulaires, sphéroïdes, organoïdes, PDX) mais aussi et surtout à partir d’échantillons de patients.. Certaines sont des méthodologies conventionnelles de biochimie et de biologie cellulaire (microscopie, gènes rapporteurs, protéines marquées…), et d’autres sont des méthodologies dédiées telles que la purification des ribosomes et des nucléoles, le fractionnement des polysomes ou l’analyse de la 2′-O-méthylation de l’ARNr par RiboMethSeq. Nous collaborons avec plusieurs équipes ayant une expertise reconnue dans le « ribosome profiling », la spectrométrie de masse et l’analyse des ribosomes par CryoEM.
Opportunités pour rejoindre l’équipe.
Si vous souhaitez rejoindre l’équipe, envoyez votre CV et une lettre de motivation à Jean-Jacques Diaz (jean-jacques.diaz@lyon.unicancer.fr).
-
Jean-Jacques DIAZ
Chef d’équipe
jean-jacques.diaz@lyon.unicancer.fr
04 78 78 28 19Cheney A – 4ème étage
Centre Léon Bérard