Plateforme RibosOMICS
Contexte
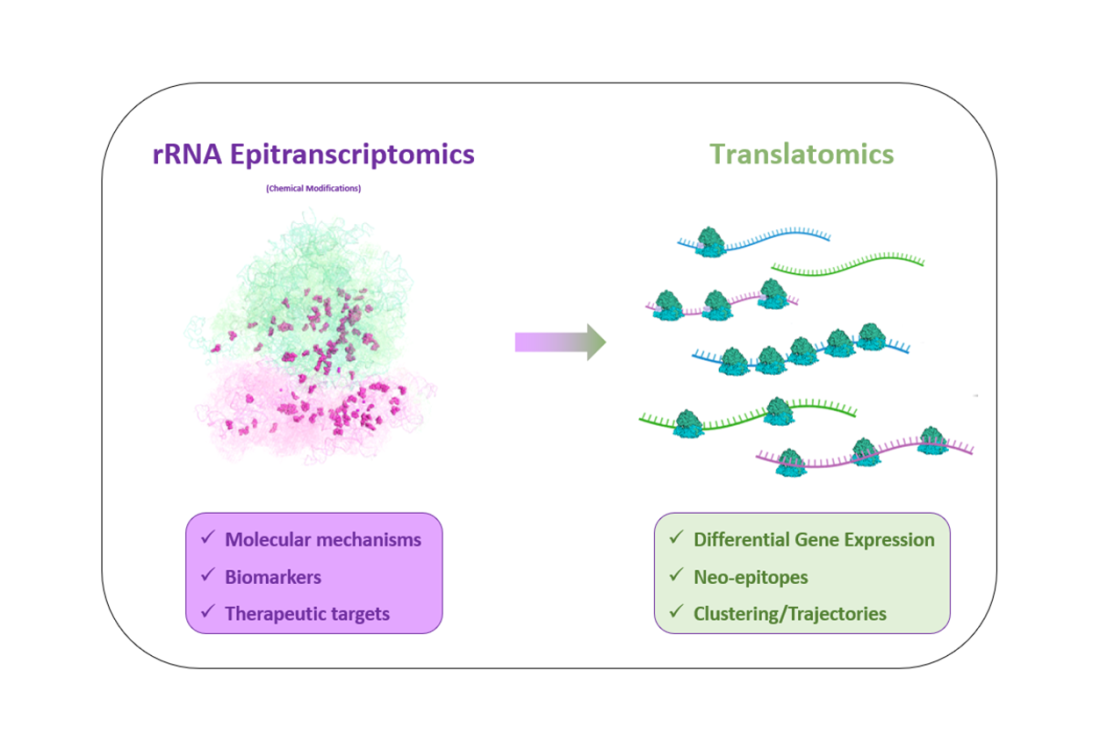
La plateforme RibosOMICS fournit une expertise complète des approches haut-débit dédiées à l’étude des différentes régulations traductionnelles telles que la translatomique (ensemble des ARNm différentiellement traduits) et l’épitranscriptomique des ARN ribosomiques (2’Omethylation et pseudouridylation).
Depuis ces 10 dernières années, il s’avère que la traduction, qui permet la synthèse des protéines à partir des ARNm par le ribosome, joue un rôle clé dans de nombreux contextes physiologiques et pathologiques, dont les cancers. Cette dernière étape de la régulation de l’expression génique contribue en effet à l’apparition de phénotypes cancéreux et à la progression des cancers, y compris à la résistance aux thérapies anti-cancers ou à la dissémination métastatique.
Aujourd’hui, comprendre le cancer à travers le prisme du contrôle traductionnel permet d’identifier de nouveaux mécanismes moléculaires dans les cancers, mais aussi de nouveaux biomarqueurs ou cibles thérapeutiques, insoupçonnés jusqu’à aujourd’hui. Parmi les principaux acteurs de la traduction, il émerge que le ribosome lui-même est un régulateur majeur de la traduction. Il s’avère que les ribosomes présentent des modifications de leurs compositions, notamment au niveau des modifications chimiques des ARN ribosomiques (ARNr), qui affectent leur activité et leur sélectivité vis-à-vis de certains ARNm.
Fonctionnement
La plateforme RibosOMICS accueille les projets du CRCL/CLB et de leurs collaborateurs, mais aussi des académiques extérieurs, et propose un support dans le design expérimental, la réalisation technique et l’analyse bio-informatique des approches suivantes :
Epitranscriptomique des ARN ribosomiques : analyse des variations du niveau des modifications chimiques des ARNr par des approches biochimiques couplées à du RNA-seq :
- RiboMethSeq (télécharger la fiche technique): analyse de la 2’O-ribose méthylation des ARNr
- Hydra(Ψ)seq (fiche technique bientôt disponible) : analyse de la pseudouridylation des ARNr
Translatomique : analyse de la traduction (ARNm différentiellement traduits et/ou évènements traductionnels) par des approches biochimiques couplées à du RNAseq ou de la RTqPCR moyen débit:
- PolySeq (fiche technique bientôt disponible) : comparaison des ARNm associés aux polysomes, c’est-à-dire aux ribosomes actifs et donc en cours de traduction. Cette approche permet d’identifier les ARNm différentiellement traduits.
- RiboSeq (fiche technique bientôt disponible) : comparaison des ARNm protégés par les ribosomes. Cette approche permet d’identifier les ARNm différentiellement traduits, et dans certaines conditions, d’identifier des défauts de traduction et des éléments régulateurs.
- RiboSTAMP (fiche technique bientôt disponible) : comparaison des ARNm édités par des ribosomes modifiés. Cette approche permet d’identifier les ARNm différentiellement traduits.
Certaines de ces approches sont réalisables en cellule unique:
- scRiboSeq (fiche technique bientôt disponible)
- scRiboSTAMP (fiche technique bientôt disponible)
Pour ce faire et simplifier les flux, la plateforme RibosOMICS assure la gestion du projet et les interactions, administratives et expérimentales, avec les autres plateformes du CRCL :
-
Management opérationnel :
Fleur BourdelaisDirection scientifique :
Virginie Marcel et Sébastien DurandVous souhaitez solliciter la plateforme RibosOMICS pour un projet.
Merci de vous référer à la procédure téléchargeable ci-dessous et de nous la transmettre à l’adresse mail suivante :
ribosomics@lyon.unicancer.fr